SeeSAR 药物设计体验
极速
几分钟内即可完成对接、分析和结构改造。
3D可视化
通过丰富的色彩来直观的展示药物结合模式。
极简
低学习成本,无论药物设计专家还是初学者都能轻松使用。
SeeSAR 核心功能

Docking Mode
探究最优结合模式、虚拟筛选和共价对接。

Molecule Editor Mode
优化分子结构,改善药物分子的类药性能。

Analyzer Mode
探究类药性参数,平衡安全性和有效性。

Inspirator Mode
探寻骨架替换、片段生长或连接获得全新结构。

Protein Mode
导入蛋白质结构,叠合结合位点。

Protein Editor Mode
突变蛋白质残基,搜索类似结合位点。

Binding Site Mode
高效预测并探索所有的可能性结合位点。

Similarity Scanner
根据化合物的分子相似性对齐您的化合物,而无需目标结构。

Space Docking Mode
Chemical Space Docking™ 工作流程,用于对超大型 Chemical Space 执行基于结构的虚拟筛选。
SeeSAR 集成组件
Chemical Space Docking™
Chemical Space Docking™ 是一种基于结构的新型虚拟筛选方法,可在超广阔的 Chemical Spaces 中筛选最有前途的候选药物。
用户可以在 SeeSAR 可视化界面中方便地启动他们的 C-S-D 工作流程。然后,HPSee 将接管计算和结果的准备工作。
通过使用共结晶配体或来自对接研究的预测结合构象作为构象生成的模板,可以增强合成子的初始锚定。这种方法可以加快计算速度,同时生成结构上与模板分子一致的构象。
Covalent Docking
共价对接在 SeeSAR 中达到了新的高度。增强版本可自动检测所提供的配体中的共价弹头,并将其转化为目标结合形式,识别 36 种最常用的弹头。
SeeSAR 在姿势生成过程中考虑了形成的共价键的灵活性,或如果确切的出口向量已知,则可以保持刚性。
此外,您可以在用户界面中方便地选择和采样任何对共价靶向有效的残基侧链。
MedChemesis - Ligand Mutation Tool
用 MedChemesis(“药物化学”和“诱变”的文字游戏),可以根据药物化学中使用的常规转化来突变配体。从配体-蛋白质复合物开始,对配体的不同转化进行采样(例如,引入甲基、用羧酸取代四唑、在芳香环系统中用碳原子取代氮原子),并为您提供有希望的建议。
这种方法的好处是只生成有趣的修改,而无需枚举每个位置的所有可能修改。例如:不对会导致分子与表面发生冲突的基团的引入进行采样。在标准硬件上,可以在几秒钟内生成 50 个建议。因此,MedChemesis 可用于创建小集合化合物,以产生后续修饰的想法,并为后续合成形成假设。
在第一次迭代中,MedChemesis 具有 290 种常用的化学反应。
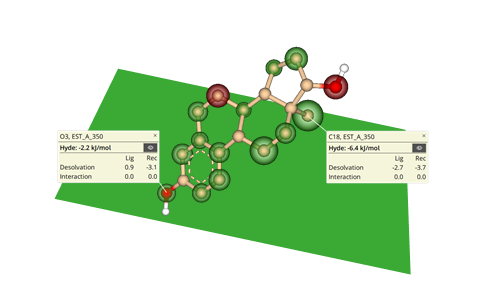
HYDE - 药物结合模式的可视化分析
HYDE 是亲和力的近似评估和可视化的工具。该系统并未针对特定靶标进行训练,而是根据基本的物理原理, 在没有任何加权参数的情况下,将氢键和去溶剂化一致性考虑得出的结果。在先导化合物优化或者其他设计任务中,您可以即时的观察到实际结果。
HYDE 由 BiosolveIT 、汉堡大学和拜尔公司合作开发,并持续改进。
ReCore-3D骨架跃迁
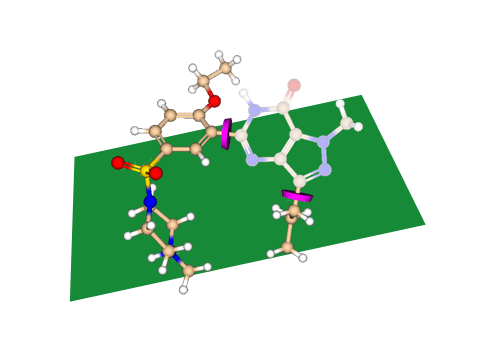
通过指定与新片段生成的键或相互作用,替换给定的骨架并生成新型的实体分子。同时为了速度,我们对片段进行了预处理,并将其集成在“index”片段库中。通过四维向量检索结果并计算匹配情况,在几秒钟内即可完成。片段库中的分子结构由 CCDC 和 PDB 蛋白库中的晶体复合物中的小分子晶体结构文件生成,具有合理的扭转键角和天然构象。
ReCore 由罗氏公司和汉堡大学的合作开发,此后被 BioSolveIT 进行广泛的扩展和功能增强。
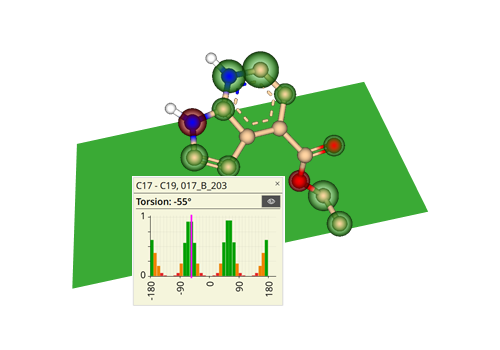
Visual torsions - 药物分子扭转角计算与分析
基于剑桥晶体数据库中的分子结构数据,对分子中各原子间的距离和扭转角进行了统计学评估。假定扭转角出现的频率和结构的能量相关,并通过不同颜色展示该二面角出现的可能性。绿色表示该二面角在天然构象中出现的频率较高,而红色表示出现的频率较低。
Visual Torsion 由 BioSolveIT 、罗氏公司和汉堡大学的合作开发。
Pocket detection - 靶标结合口袋的三维展示
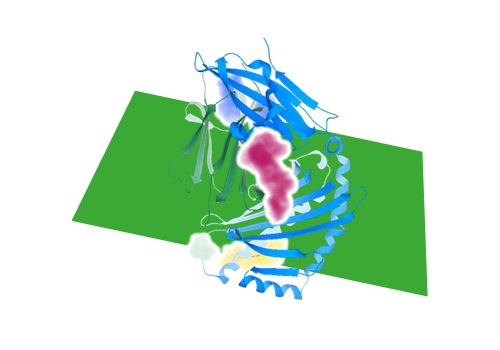
高效预测蛋白质的可结合位点,并将结合位点进行三维的展示。基于启发式模型,Pocket detection在 3D 网格上评估可能的结合部位形状,同时考虑了全局氢键、亲脂性以及溶剂可及表面 (SAS),可有效地预测药物的结合部位。
Pocket detection 是 DoGSiteScorer 的一部分,由BiosolveIT、汉堡大学和默克公司合作开发的。
FlexX – 基于片段生长的对接算法
FlexX可高效的完成分子对接。这种先进的算法将配体分成若干片段,这些片段被放置在口袋中的多个位置,并使用简单但非常快速的预评分方案进行评分。从放置的 n 种方法中,进一步扩展,进行片段连接,中间结构之间相互评分。得分最高的在整个过程中被保留下来,并将其交付给用户。当化合物缺少极性基团时,“单交互扫描”(SIS) 也能找到解决方案。

FlexS - 基于配体的相似性搜索
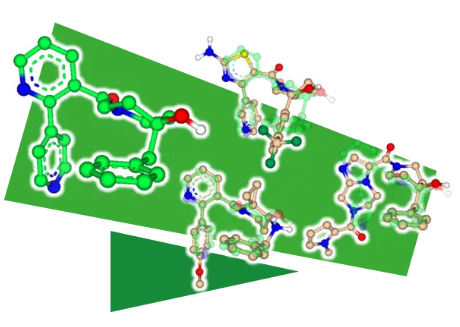
FlexS 是一种用于预测配体比对的计算机程序。对于一对给定的配体,FlexS 预测其中一个配体相对于另一个配体的构象和方向。无需目标 3D 结构,用户就可以将 FlexS 用作相似性扫描仪模式的一部分,对分子组进行虚拟筛选,以发现与参考配体相似的化合物。FlexS 考虑了分子的形状和分子特征,可用于支架跳跃和生成结合模式假设。
FastGrow — 快如闪电的口袋探索
FastGrow 使用户能够以惊人的速度快速搜索配体修饰或延伸,以补充靶结构的未占用结合腔。这种新颖的工具实现了交互式探索性生长,并将对生长过程的控制权牢牢地交还给用户。此外,用户可以应用药效团约束,以根据自己的需要微调搜索。
FastGrow 由汉堡大学与 Servier Paris 和 BioSolveIT 联合开发,已经在 艾伯维 取得了初步成功。它已经在各种场景中的真实片段增长/替换上得到了彻底的验证。
Optibrium - ADMET 属性预测
理化性质是决定一种化合物是否可能成为候选药物的重要因素。SeeSAR不仅提供了配体和受体之间易于理解的亲和力和物理特性等参数,其集成的 Optibrium 模块还可以进行ADMET性能预测,使用户可以在药物发现的早期阶段评估化合物的ADMET性能,提前预知其毒性和药代问题,从而进行规避。
SeeSAR 五大核心优势

案例分享
- Biasini等人以朊病毒蛋白 (PrP)折叠中间体为靶点,进行虚拟筛选,发现了四种苗头化合物。在体外实验中,证实它们都能够通过促进蛋白质降解来选择性地降低蛋白质的负载,从而为通过作用于其折叠途径来调节靶蛋白的水平这种全新的作用机制铺平了道路。
- Pharmacological inactivation of the prion protein by targeting a folding intermediate.
- https://www.nature.com/articles/s42003-020-01585-x
- 人类免疫缺陷病毒 1 型 (HIV-1) 衣壳抑制剂 PF-74 的代谢稳定性差是其临床应用发展的主要问题。为了提高代谢稳定性,Cocklin等人介绍了一种新颖的多步骤计算驱动工作流程以改进代谢稳定性。与起始化合物相比,成功将半衰期增加200倍。
- Rapid Optimization of the Metabolic Stability of a Human Immunodeficiency Virus Type-1 Capsid Inhibitor Using a Multistep Computational Workflow
- https://pubs.acs.org/doi/10.1021/acs.jmedchem.0c01810
- 在本出版物中,Günther 等人报道了含有4-酰基吡咯单元的新型BET-BRD7/9抑制剂的设计。使用 SeeSAR 进行分子对接研究以预测纳摩尔化合物 11 的结合模式并阐明末端基团中氧原子的有益作用。
- 4-Acyl Pyrroles as Dual BET-BRD7/9 Bromodomain Inhibitors Address BETi Insensitive Human Cancer Cell Lines
- https://pubs.acs.org/doi/10.1021/acs.jmedchem.0c00478






